Na divisão celular, especificamente na meiose, se cada par de alelos estiver em um diferente par de cromossomos, por exemplo RrIi x rrii, teremos como resultado esperado a produção de quatro classes fenotípicas na prole em uma proporção de 1:1:1:1. Com esse arranjo não-ligado de alelos, qualquer um dos membros de um par podem combinar-se aleatoriamente com qualquer um dos membros do outro par, resultando na produção de quatro tipos de gametas, RI, Ri, rI, ri, em igual número pelo individuo duplo heterozigoto.
Com o avanço da genética, tornou-se claro que o número de genes por espécie excede o número de pares de cromossomos. Como exemplo, temos a Drosophila melonogaster, uma mosca de frutas que tem cerca de 5.000 genes. Como esta espécie tem apenas quatro pares de cromossomos, cada um dos cromossomos tem de transportar muitos genes, que é a expectativa para todos os organismos eucarióticos.
Todos os genes que são transportados em um par de autossomos constituem um grupo de ligação, e o esperado é que fossem herdados em conjunto, se não fosse o crossing-over (evento onde há troca de informação genética entres as cromátides homólogas). Assim seria esperado que a quantidade de grupos de ligação para qualquer organismo fosse igual ao número haplóide de cromossomos (essa expectativa terá que ser emendada quando se considerar os cromossomos sexuais).
É interessante que a ligação foi prevista muito antes de ser realmente demonstrada. Apenas três anos após a descoberta do trabalho pioneiro de Mendel, Sutton (1903) sugeriu que cada cromossomo tem mais que um alelo e que os alelos representados por um cromossomo qualquer devem ser herdados juntos. Porém, Sutton não foi capaz de sustentar experimentalmente sua hipótese e apenas poucos anos depois, Bateson e Punnett (1905-1908) chegaram a ter dados para realizar o experimento, mas ainda sem reconhecer que estavam tratando de pares de alelos localizados no mesmo cromossomo (genes em ligação).
1.2 Notação de Cruzamentos com Ligação
Analisando os cruzamentos com genes em ligação, deve-se saber os genótipos dos indivíduos cruzados e também a disposição dos genes no cromossomos. Considere um cruzamento entre um indivíduo homozigoto para alelos dominantes em dois loci ligados e outro indivíduo homozigoto para alelos recessivos nesse loci(AABB X aabb). Desta forma é necessário escrever os alelos específicos á medida da disposição em cada um dos cromossomos homólogos.
Nessa notação, cada linha representa um dos dois cromossomos homólogos. Herdando um cromossomo de cada genitor, a prole F1:
Fonte: Pierce (2011)
É importante lembrar que dois alelos em um locus são sempre situados em cromossomos homólogos diferentes, isso significa que devem estar em lados opostos da linha. Também é importante sempre ter a mesma ordem dos genes em ambos os lados da linha, pois os genes que estiverem sobrepostos serão alélicos (no mesmo locus).
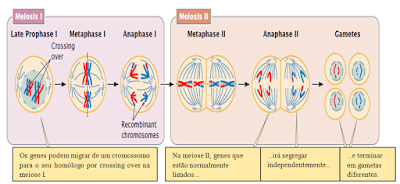
2. Permuta gênica
A permutação ocorre na meiose I, esta fase se divide em Prófase I, Prometáfase I, Metáfase I, Anáfase I e Telófase I. A Prófase I se divide em Pré leptóteno, Leptóteno, Zigóteno, Paquíteno, Diplóteno e Diacinese.
Durante o paquíteno, os cromossomos se condensam e o emparelhamento dos cromossomos homólogos se completa. Porém o mais importante deste período é a troca de informação genética entre as cromátides homólogas, fenômeno que leva o nome de permutação (recombinação gênica ou em inglês crossing-over), que tem uma importante significativa por ser fonte de variabilidade genética, como mostra a figura a seguir.
Figura 1. O crossing-over ocorre na meiose e é responsável pela recombinação. Fonte: Adaptado de Pierce (2011).
O paquíteno é um processo longo, levando dias de duração, diferente de outras fases da prófase I que levam apenas algumas horas para ocorrer completamente, como por exemplo, leptóteno e zigóteno.
No paquíteno o núcleo aparenta conter um número haplóide de cromossomos, porém cada uma das unidades visíveis é composta de dois cromossomos independentes e intimamente emparelhados. Isto faz com que cada um dos 23 pares de cromossomos receba o nome de bivalente. Como cada conjunto é composto por quatro cromátides, chama-se também de tétrade, como mostra a imagem a seguir.
Figura 2. Esquema demonstrando a tétrade, formação de quiasma e a separação dos cromossomos.Fonte: De Robertis (2010).
As duas cromátides irmãs de cada cromossomos são ligados pelo centrômero e por isso em um bivalente ou tétrade existem dois centrômeros, um por cromossomos. Como na mitose, cada centrômero contem dois cinetócoros, um por cada cromátide irmã. Porém, até na finalização da mitose I os cinetócoros irmãos se comportam como apenas uma unidade. Durante o bivalente, no CS (complexo sinaptonêmico), uma estrutura complexa, aparece uma sucessão de nódulos densos de cerca de 100nm de diâmetro, denominados nódulos de recombinação. Seu número e suas localizações coincidem com os sítios de recombinação gênica, o que indica que ao nível deles acontece o intercâmbio de segmentos de DNA entre as cromátides homólogas.
Para que a permuta ocorra, as moléculas de DNA das cromátides homólogas devem estar situadas a uma distância de aproximadamente 1 nm no componente central do CS. Acredita-se que esse contato ocorra a nível dos filamentos transversais que unem os componentes laterais, pelos quais as sequências de nucleotídeos homólogos se buscariam, fatos imprescindível para que aconteça a troca de segmentos de DNA. O nódulo de recombinação poderia ser considerado a expressão morfológica dessa troca. Além disso, o nódulo seria um complexo multiprotéico que reúne as cromátides paternas e maternas e produz os cortes e as emendas necessários para a recombinação.
Entre as proteínas que participam da recombinação, se destaca a Rad51 (de radiation sensitive), cujo o papel é essencial para que ocorra a formação de uma tripla hélice transitória no começo da recombinação gênica, como ilustrado a seguir.
Figura 3. Formação de uma tripla hélice transitória no começo da recombinação genética.Fonte: De Robertis (2010).
2.1 Permuta gênica com genes ligados
No caso de genes proximamente ligados, não ocorre crossing over em todas as meioses, portanto, nas meioses que não há crossing, resultam em gametas não recombinantes.A ligação e o crossing over podem ser vistos como um processo que tem efeitos opostos; a ligação mantém juntos determinados genes e o crossing over os mistura. Na meiose que há um único crossing over, onde afeta apenas duas das quatro cromátides, metade dos gametas são recombinantes e outra metade não são recombinantes. Assim, a porcentagem de gametas recombinantes é sempre metade da porcentagem de meioses nas quais ocorre o crossing over. Mesmo que o crossing over entre dois genes ocorra em cada meiose, apenas 50% dos gametas resultantes serão recombinantes. Assim a freqüência de gametas recombinantes é sempre a metade da freqüência de crossing over, e a proporção máxima de gametas recombinantes é 50%.
Figura 4. Um único crossing over produz metade dos gametas não recombinantes e a metade de gametas recombinantes. Fonte: Adaptado de Pierce (2011).
2.2 Calculando a Frequência de Permuta
A Frequência de permuta é a porcentagem de prole recombinante produzida em um cruzamento, e pode ser calculada da seguinte forma:
2.3 Aclopamento e Repulsão
O arranjo dos alelos em cromossomos homólogos é critico em determinar o resultado do cruzamento para genes ligados. Por exemplo, considere a herança de dois genes em mosca-varejeira australiana, Lucilia cuprina. Nessa espécie, um locus determina a cor do tórax: púrpura (p) é recessiva para o tórax verde normal (A). Um segundo determina a cor da pupa: preta (b) é recessiva em relação à pupa marrom normal (B). Esses loci estão situados próximos no segundo cromossomo.Suponha que tentamos cruzar uma mosca que é heterozigota em ambos os locis com uma mosca que é homozigota recessiva em ambos. Como esses genes são ligados, existem dois arranjos possíveis nos cromossomos da prole da mosca heterozigota. Os alelos dominantes para tórax verde (A) e pupa marrom (B) devem estar no mesmo cromossomo, e os alelos recessivos para tórax púrpura (a) e pupa preta (b) devem residir no outro cromossomo homólogo.
O arranjo, no qual os alelos tipo selvagem são encontrados em um cromossomo e os alelos mutantes são encontrados no outro cromossomo, é chamado de acoplamento ou configuração cis. Alternadamente, um cromossomo pode ter os dois alelos para tórax verde (A) e pupa preta (b), e o outro cromossomo levaria os alelos para tórax púrpura (a) e pupa marrom (B), neste caso, no qual cada cromossomo possui um tipo selvagem e um alelo mutante, é chamado de repulsão ou configuração trans.
Figura 5. Demonstração de configuração cis e trans em cromossomos homólogos. Fonte:Dos Santos (2017).
2.4 Teste para Segregação Independente
Em alguns cruzamentos os genes são claramente ligados porque há mais prole não recombinante do que prole recombinante. Em outros cruzamentos, a diferença entre a segregação independente e a ligação não é tão óbvia. Por exemplo,, suponha que tenhamos feito um cruzamento-teste para dois pares de genes, tais como AaBb X AA bb, e observado os seguintes resultados da prole: 54 AaBb, 56 aabb, 42 Aabb e 48 aaBb. Esse resultado tem a proporção 1:1:1:1 que esperaríamos se A e B segregarem independentemente? Não exatamente, mas bem próxima. Talvez esses genes segreguem independentemente e ao acaso tenham gerado leves desvios entre a proporção 1:1:1:1 esperada e os números observados. Alternativamente, os genes podem estar ligados, com provável crossing over ocorrendo entre eles, e dessa forma o numero de não recombinantes é apenas levemente maior do que o numero de recombinantes.
Nos deparamos então com uma questão: Como distinguir o papel do acaso e o papel da ligação em produzir desvios dos resultados esperados com segregação independente? Se a herança dos alelos em um locus for independente da herança de alelos em um segundo locus, então os genes estão segregados independentemente. Porém, se a herança dos alelos em um locus não for independente da herança dos alelos em um segundo locus, então os genes provavelmente estão ligados.
Um modo de testar a distribuição independente é calcular a probabilidade esperada de cada tipo de prole, supondo segregação independente, e então usar o teste de qui-quadrado da verossimilhança para avaliar se os números observados desviam-se significadamente dos números esperados. Com a distribuição independente, esperamos ¼ de cada fenótipo, essa probabilidade esperada de cada genótipo é baseada na regra da multiplicação da probabilidade, por exemplo, se a probabilidade de Aa é ½ e a probabilidade de Bb é ½ , então a probabilidade de AaBb é ½ X ½ = ¼. Vamos fazer duas suposições: (1) a probabilidade de cada genótipo de um só locus é ½ e (2) os genótipos nos dois loci são herdados independentemente (½ X ½ = ¼).
Um problema com esse enfoque é que um teste significativo de qui-quadrado pode resultar de uma violação de uma das suposições. Se os genes estiverem ligados, então as heranças dos genótipos nos dois loci não são independente (suposição 2), e teremos um desvio significativo entre os números observados e esperados. Mas também podemos ter um desvio significativo se a probabilidade de cada genótipo de um locus não é ½ (suposição 1), mesmo quando os genótipos estão segregados independentemente.Podemos testar ambas as suposições conduzindo uma série de testes de qui-quadrado, primeiro testando a herança dos genótipos e em cada locus separadamente (suposição 1) e, então testando a distribuição independente (suposição 2). Entretanto, um método mais rápido é testar a independência em genótipos com um teste de qui-quadrado de independência.
O teste do qui-quadrado de independência nos permite avaliar se a segregação de alelos em um locus é independente da segregação de alelos em outro locus, sem fazer nenhuma suposição sobre a probabilidade de genótipos em um só locus.
Para fazermos esse teste, devemos construir um quadro de contingência dos números observados da prole, similar ao quadro de Punnett, exceto que nesse pomos os genótipos que resultam da segregação de alelos em um locus na parte de cima e os genótipos que resultam da segregação de alelos no outro locus ao longo da lateral. Em seguida, computamos o total para cada fila, o total para cada coluna e o total geral ( a soma do total das fileiras ou a soma do total da colunas, que devem ser iguais). Esses valores serão usados para computar os valores esperados para o teste do qui-quadrado da independência.
Com a suposição de que os alelos nos dois loci segregam-se independentemente, o número esperado para cada célula do quandro pode ser computado usando a seguinte fórmula:
Agora calculamos o valor do qui-quadrado usando a mesma fórmula que usamos para o teste de qui-quadrado de verossimilhança:
Para determinar a probabilidade associada ao valor de qui-quadrado, precisamos dos graus de liberdade, que são o número de modos pelos quais as classes observadas estão livres para variar dos valores esperados.
Em geral, para o teste do qui-quadrado de independência, os graus de liberdade são iguais ao número de filas em um quadro de contigência menos 1 multiplicado pelo número de colunas no quadro menos 1, ou:
df = (número de filas - 1) × (número de colunas - 1)
Depois de obter o valor de qui-quadrado e os graus de liberdade, podemos obter a probabilidade de um quadro de qui-quadrado (Tabela 1). Os graus de liberdade são dados na coluna da esquerda do quadro e as probabilidades são dadas em cima; no corpo da tabela estão os valores de qui-quadrado associados a essas probabilidades. Primeiro ache a coluna dos graus apropriados de liberdade. Encontre onde o qui-quadrado está entre os valores nesta coluna. Os valores teóricos de qui-quadrado aumentam da esquerda para a direita e as probabilidades diminuem da esquerda para a direita.
Fonte: Pierce (2011)
A maioria dos cientista usam o nível de probabilidade de 0,05 como valor limítrofe: se a probabilidade de o acaso ser responsável pelo desvio é superior ou igual a 0,05, eles aceitam que o acaso pode ser responsável pelo desvio entre o observado e os valores esperados. Quando a probabilidade é menos do que 0,05, os cientistas supõem que o acaso não é responsável e existe uma diferença significativa, o que significa no caso que os genes não estão segregando independentemente, ou seja, estão em ligação.
3. Mapeamento gênico
Segundo Morgan, as distâncias físicas entre os genes estão relacionadas com as taxas de recombinação, formulando uma hipótese onde diz que os eventos de crossing over ocorrem mais ou menos ao acaso para cima ou para baixo no cromossomo, e que dois genes que estão distantes têm mais probabilidade de sofrer crossing over do que dois genes que estão bem próximos. Propuseram então que as frequências de recombinação podem ser um modo para determinar a ordem nos genes ao longo de um cromossomo.
As distâncias em um mapa genético são medidas em unidades mapa (m.u.), e uma unidade mapa equivale a 1% de permuta. As unidades mapa também são chamadas de centimorgans (cM). As distâncias genéticas medidas com a taxa de recombinação são aproximadamente aditivas, isso significa que se a distância do gene A ao gene B é de 5 m.u., a distância do gene B ao gene C é de 10 m.u. e a distância do gene A ao gene C é de 15 m.u., então o gene B deve estar localizado entre os genes A e C. Com base nesse exemplo podemos desenhar um mapa genético simples para os genes A, B e C:
Fonte: Pierce (2011)
Seria possível desenhar também o mapa com o gene C à esquerda e o gene A à direita:
Fonte: Pierce (2011)
Ambos os mapas estão corretos e equivalentes e fazendo uma série de cruzamentos entre pares de genes, é possível construir mapas genéticos mostrando os arranjos de ligação de vários genes.
É importante destacar dois pontos em relação a construção de mapas genéticos pela freqüência de recombinação. Primeiro, não podemos distinguir genes em cromossomos diferentes e genes situados bem distantes no mesmo cromossomo. O segundo ponto é que o cruzamento-teste para dois genes que estão relativamente distantes no mesmo cromossomo tende a subestimar a verdadeira distância física, pois o cruzamento não revela os crossing overs duplos que podem ocorrer entre os dois genes.
Figura 6. Um crossover duplo de dois filamentos entre dois genes ligados produz apenas gametas não recombinantes. Fonte:Adaptado de Pierce (2011).
3.1 Cruzamento-teste de Três Pontos
Os mapas genéticos podem ser construidos a partir de uma serie de cruzamentos-teste para pares de genes, mas esse enfoque não é particularmente eficiente, pois vários cruzamentos de dois pontos devem ser feitos para estabelecer a ordem dos genes, e porque os crossovers duplos são perdidos.
Com o cruzamento de três pontos, a ordem dos genes pode ser estabelecida em um único grupo de prole e alguns crossovers duplos podem geralmente ser detectados, dando distâncias de mapa precisas, por isso se torna uma técnica mais eficiente de mapeamento.
Considere o que acontece quando ocorre um crossing over entre três genes ligados hipotéticos. Veja a figura:
Figura 7. Três tipos de crossovers podem ocorrer entre três loci ligados. Fonte:Adaptado de Pierce (2011).
A figura ilustra um par de cromossomos homólogos de um indivíduo que é heterozigoto em três loci (Aa Bb Cc). Note que os genes estão em configuração de acoplamento (cis), o que significa que todos os alelos dominantes estão em um cromossomo (A B C) e todos os alelos recessivos estão em outro cromossomo (a b c). Três tipos de eventos de crossover podem ocorrer entre esses três genes: dois tipos de crossover únicos e um crossover duplo, como na figura a cima. Em cada tipo de crossover, dois dos cromossomos resultantes são recombinantes e dois não recombinantes.
Note que, nos cromossomos recombinates resultantes de crossover duplo, os dois alelos mais externos são os mesmos que nos não recombinantes, mas o alelo médio é diferente. Esse resultado nos fornece um importante indício sobre a ordem dos genes. Na prole que resulta de um crossover duplo, apenas o alelo do meio deve diferir dos alelos presentes na prole não recombinante.
Referências
BURNS, George W.; BOTTINO, Paul J. Genética. 6.ed.rev. Tradução João Paulo de Campos et al.Rio de janeiro, RJ: Guanabara Koogan,1991.
DE ROBERTIS, Eduardo M. F. Bases da biologia celular e molecular. 4.ed.rev. Tradução Antonio Francisco Dieb Paulo.Rio de janeiro: Guanabara Koogan, 2006.
DOS SANTOS,Vanessa S.Linkage. Mundo Educação. Copyright © 2017. Disponivel em:
PEIRCE, Benjamin A. Genética: um enfoque conceitual. 3.ed. Tradução Paulo A. Motta. Rio de Janeiro: Guanabara Koogan, 2011.












Nenhum comentário:
Postar um comentário